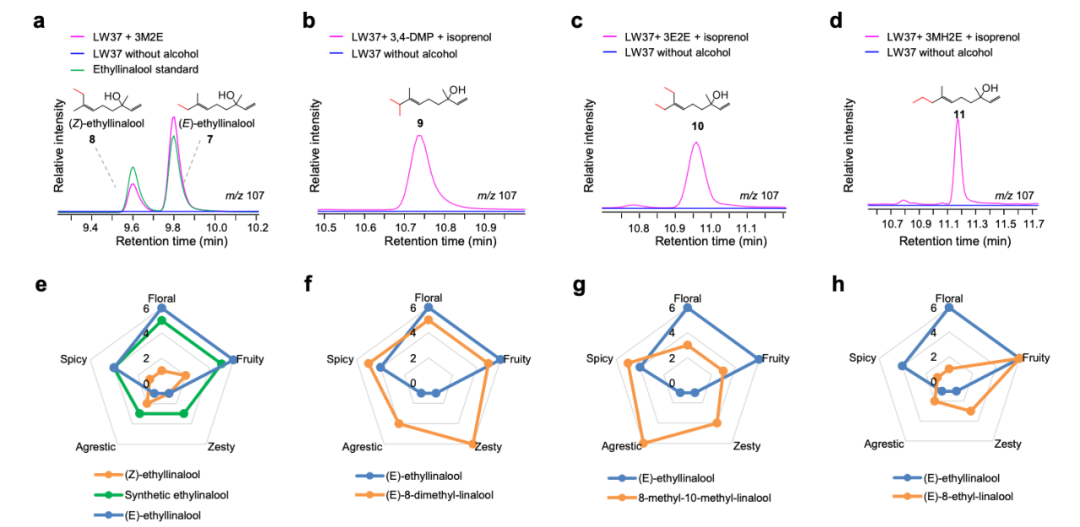
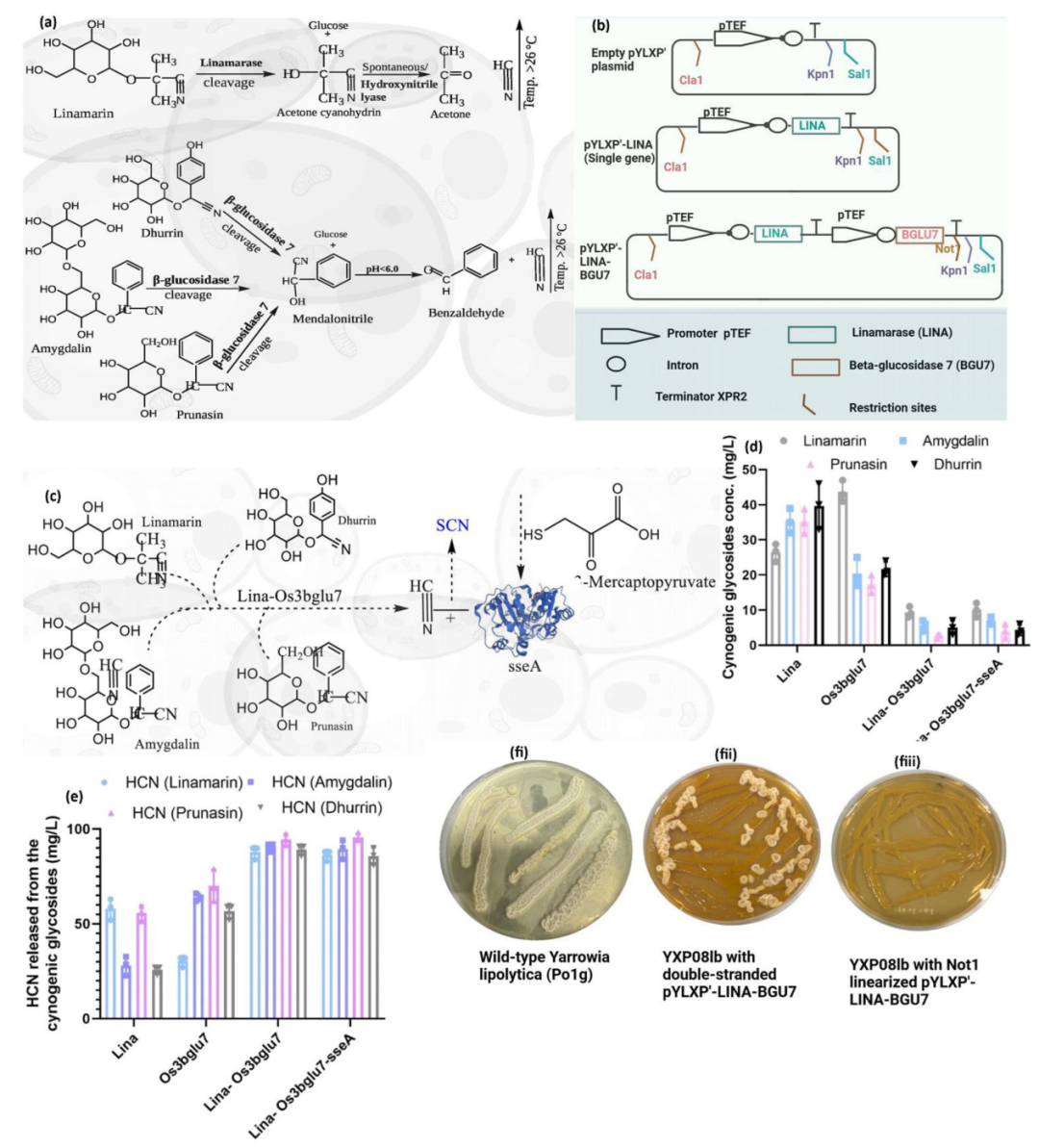
近年来,科学家们尝试通过“遗传密码扩展”技术,将非天然氨基酸(ncAAs)引入蛋白质结构中,为其赋予光响应、化学修饰、药物结合等多种新功能,这一技术正推动蛋白质工程进入“设计蛋白”的新阶段。然而实现这一目标的关键瓶颈是——如何精准、高效地将这些特殊氨基酸插入蛋白质中?
吡咯赖氨酰-tRNA 合成酶(PyIRS)是目前最常用的工具之一,它能够识别并激活非天然氨基酸,将其连接到对应的 tRNA 上,从而在蛋白质翻译过程中插入目标位置。然而,PylRS 虽然具备一定识别能力,但面对不同非天然氨基酸时效率不一,导致含有非天然氨基酸的蛋白质表达量远低于野生型蛋白质,严重限制了其应用广度,传统的定向进化策略则需要针对每个目标非天然氨基酸进行多轮筛选,耗时费力。
为了突破这一关键技术瓶颈,近日,浙江大学于浩然等人在 Nature Communications 上发表一篇题为“Machine learning-guided evolution of pyrrolysyl-tRNA synthetase for improved in“的研究,该团队报道了一种以机器学习为基础的新型酶工程策略,可大幅提升 PylRS 酶对非天然氨基酸的识别与掺入效率,为蛋白质工程功能扩展供了强大工具。
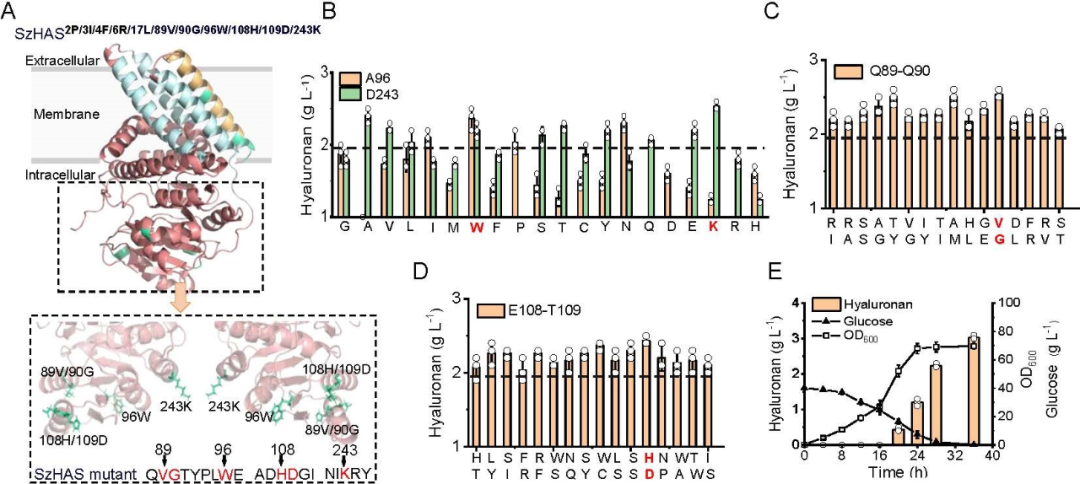
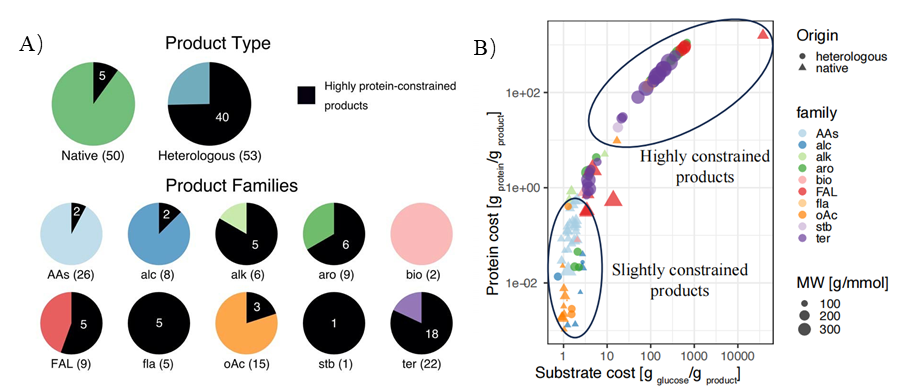
研究人员首先基于已有文献整理出 PylRS 酶上 12 个可能影响效率的突变位点,并基于这些位点构建了 4096 种不同的突变组合。
随后,他们使用了一种名为 FFT-PLSR 的机器学习模型,结合已有实验数据,对所有组合进行模拟预测,筛选出潜力最大的突变版本。最终,他们找到了第一个性能优异的变体——Com1-IFRS,其掺入非天然氨基酸的能力比原始酶高出 11 倍。接着,团队进一步引入深度学习模型,在更广泛的序列空间中搜索潜在突变位点,并结合前一轮筛选数据进行再训练,最终获得性能更优的变体 Com2-IFRS,其识别效率和蛋白表达量提升了 30 倍以上,基本接近天然氨基酸系统的表现。
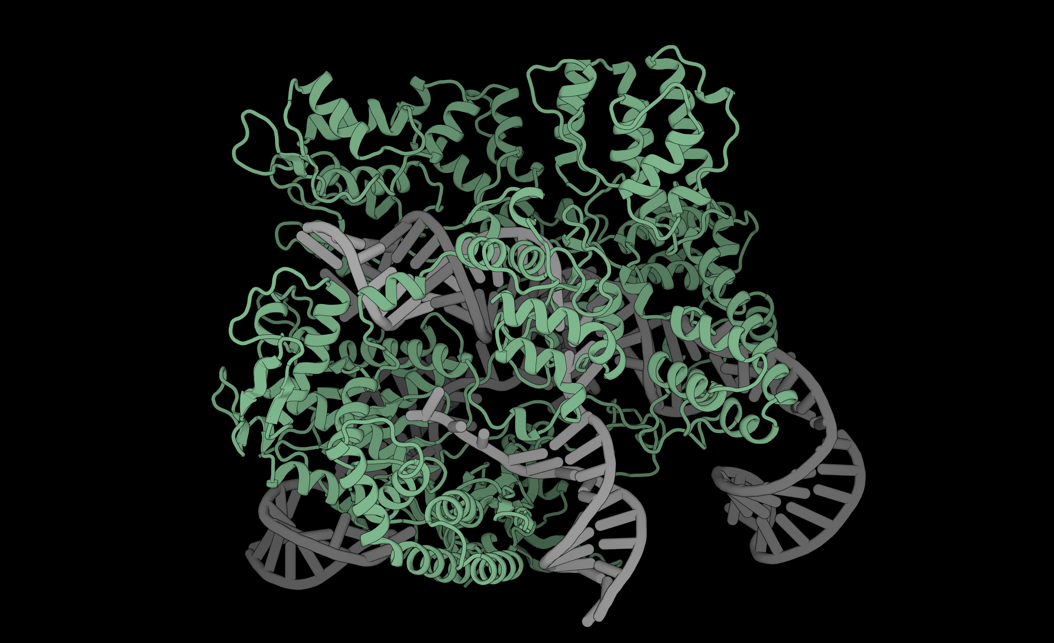
令人意外的是,研究人员这些提高效率的突变并不是发生在 PylRS 用于识别底物(ncAA)的核心区域,而是集中在 tRNA 结合区域(TBD),换句话说,它们并不会改变酶识别哪种氨基酸,而是能帮助酶更加快速稳定的结合 tRNA,从而提升整个催化过程的稳定性。因此,这组突变可以像插件一样模块化迁移到各种不同的 PylRS 变体中,通用性极强。
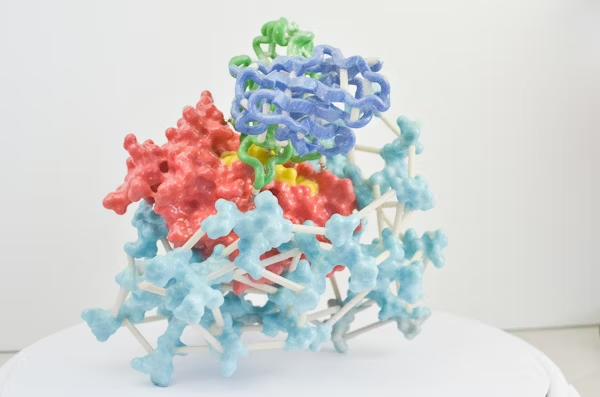
 图 | 关键突变集中发生在 PylRS 的 tRNA 结合域,大幅提高通用性
图 | 关键突变集中发生在 PylRS 的 tRNA 结合域,大幅提高通用性
为验证这种“插件”的通用性,团队将 Com1 和 Com2 突变分别“嫁接”到 7 种识别不同非天然氨基酸的 PylRS 变体中,包括 3-氟苯丙氨酸(3FF)、3-溴酪氨酸(3BrY)、带保护基的赖氨酸类(BocK、AlocK)、色氨酸衍生物等。
结果表明,不管是哪个变体、识别哪类非天然氨基酸,只要加入 Com1 或 Com2 突变,其识别效率和蛋白表达能力几乎全部显著提升。
以 3FF 为例,Com2 变体驱动的绿色荧光蛋白表达量比未突变版本提升超过 101.9 倍,几乎与天然氨基酸掺入效率持平。对其他底物的表现也同样惊艳,3BrY 提升近 120 倍,BocK 和 AlocK 提升 40.2 倍和 32.7 倍,原本表达效率偏低的色氨酸类底物,在 Com2 加持下提升幅度也超 50 倍。这意味着,只需一套优化突变插件,就可以大幅提升多种底物掺入效率,为系统快速适配各种新型非天然氨基酸提供了现实路径。
在实际应用层面,该优化策略也表现良好。研究人员在将 Com2 系统用于合成带有 3-甲基组氨酸修饰的肌红蛋白时,与原始系统相比,Com2 系统驱动下该蛋白表达量提高了 6 倍,其酶催化能力也增强近 8 倍。
 图 | Com2 系统驱动下,含 3MetH 修饰的肌红蛋白表达量显著提升
图 | Com2 系统驱动下,含 3MetH 修饰的肌红蛋白表达量显著提升
那么,这些突变为什么能起作用?为了揭示其机制,团队借助 AlphaFold3 构建了 PylRS 的结构预测模型,并进行分子动力学模拟。他们发现,在 Com2 突变体中,底物和 tRNA 之间的距离更短,形成了多个新的氢键,这些结构调整让整个底物装配过程更高效,正是导致掺入效率大幅提升的物理基础。
更进一步,研究团队还验证了这些突变在多点掺入(即在蛋白中同时插入多个非天然氨基酸)的表现。他们构建了一系列带有 1 至 5 个位点的终止密码子(TAG)的绿色荧光蛋白,并测试 Com1 和 Com2 系统能否高效完成掺入。结果表明,即便是 5 个位点同时插入,Com2 突变体依然能保持稳定高效的表达能力,显示出极强的扩容能力,这使得该系统可广泛应用于构建多点修饰蛋白、交联蛋白、光控开关蛋白等复杂设计。
总体来看,这项研究创新性地将机器学习引入非天然氨基酸掺入酶的设计与进化中,构建了一条以“组合突变预测、深度学习筛选、模型重训练”为核心的优化路径,显著提高了 PylRS 系统对多样性底物的识别能力。未来,相信随着训练数据的进一步积累与算法模型的不断优化,该方法有望拓展至更广泛的酶工程任务中,为合成生物学、蛋白质药物设计等领域提供更加高效与可控的工具。
参考文献:
1. Zhang, Q., Jiang, L., Niu, Y. et al. Machine learning-guided evolution of pyrrolysyl-tRNA synthetase for improved incorporation efficiency of diverse noncanonical amino acids. Nat Commun 16, 6648 (2025). https://doi.org/10.1038/s41467-025-61952-2
免责声明:本文旨在传递合成生物学最新讯息,不代表平台立场,不构成任何投资意见和建议,以官方/公司公告为准。本文也不是治疗方案推荐,如需获得治疗方案指导,请前往正规医院就诊。
























































































































































































安各洛(深圳)生物科技有限公司 版权所有