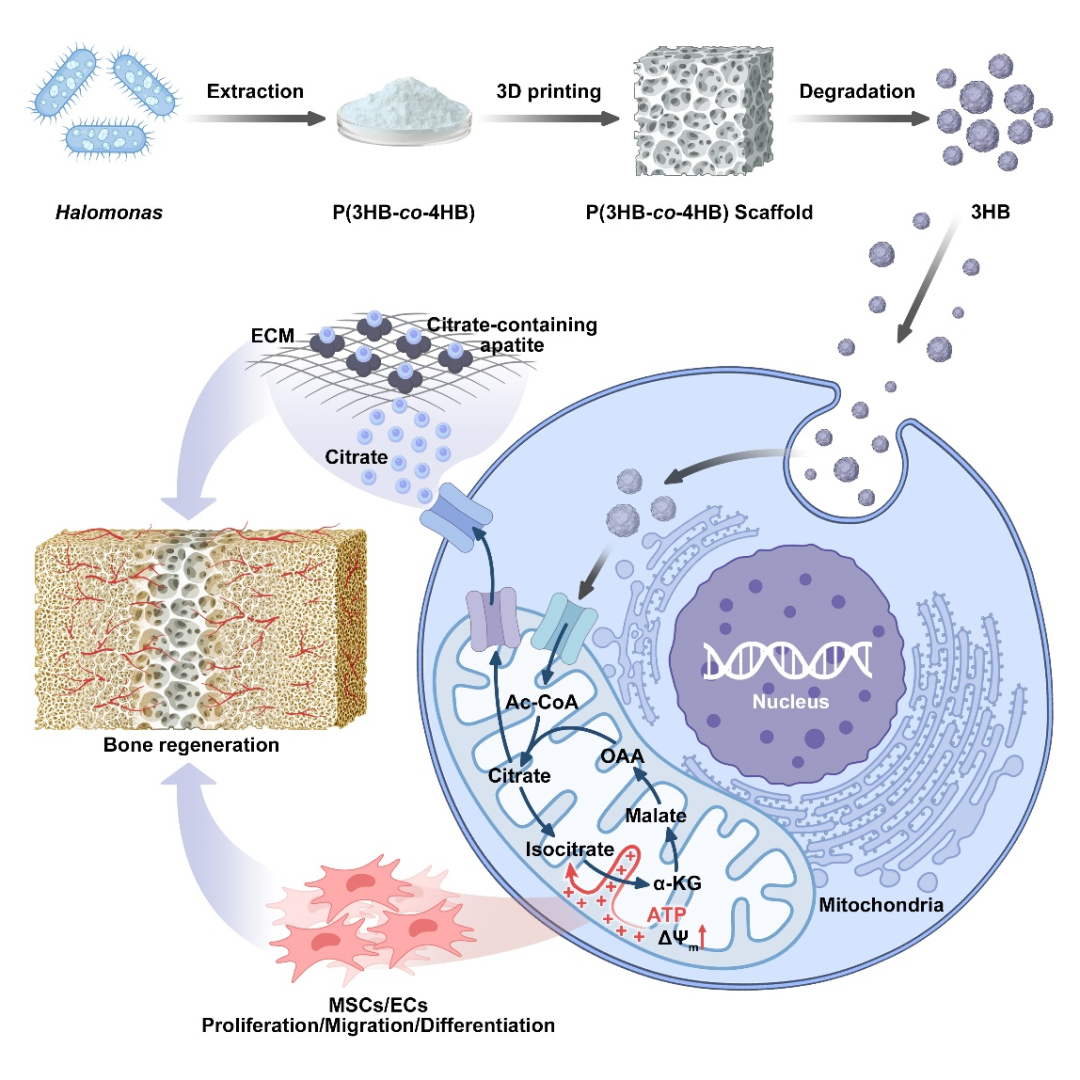
基因编辑技术有望解决农业、生物技术和人类健康领域的根本性挑战。源自微生物的 CRISPR 基因编辑器种类繁多、功能强大,但在临床应用时,仍存在脱靶效应,并可能引发免疫系统不良反应,限制了其更广泛的治疗应用。
2024 年 4 月,总部位于加州伯克利的 AI 蛋白质设计公司 Profluent Bio 发布了首个利用 AI 设计的基因编辑器 OpenCRISPR-1,并成功编辑了人类基因组。Profluent 于 2023 年以 900 万美元的种子轮融资启动,并于 2024 年获得了额外的 3500 万美元融资。
近日,相关成果正式发表在 Nature 期刊。该论文在之前发表的预印本的基础上进行了扩展,并提出了几项新发现,用于增强 AI 在蛋白质设计中的有效性。
AI 生成具有突破性新颖度和多样性的序列:基于大规模生物序列训练的大型语言模型(LLMs)能够生成涵盖多家族 的 CRISPR-Cas 序列,其与已知天然蛋白存在数百个突变差异,这种多样性通过人工设计无法实现。
LLMs 在 CRISPR-Cas 设计中的可行性验证:Profluent 平台设计的 Cas 蛋白功能表现显著优于传统人工设计及其他机器学习蛋白工程方法,展现出卓越的工程化能力。
OpenCRISPR-1 全基因组脱靶分析:通过无偏倚的全基因组特异性检测,OpenCRISPR-1 展现出显著降低的脱靶切割事件。
OpenCRISPR-1具有更低免疫原性潜力:定量 iELISA 检测表明,所有 AI 生成的 Cas 蛋白相较 SpCas9 均呈现更低免疫原性,这预示机器学习设计的“自然界前所未有”蛋白相较于病原体来源的基因组编辑器(如 SpCas9)可能具有更优的免疫兼容性。
这些研究成果验证了 Profluent 的大型语言模型可用于生成定制基因编辑器,其性能可匹敌甚至超越天然 CRISPR 系统,并有望扩展基因编辑能力并加速治疗药物的研发。

为了设计性能可匹敌甚至超越天然 CRISPR 系统,团队投入了大量时间进行详尽的数据挖掘,构建了 CRISPR-Cas Atlas,这是一个通过筛选 26.2 万亿个组装微生物基因组碱基而构建的资源库。其中包括 Cas 蛋白、CRISPR 阵列、tracrRNA 和 PAM 序列。
相较于 CRISPRCasDB、CasPDB 等精选数据库,以及全球最大蛋白质数据库 UniProt,上述数据库展现出更丰富的天然多样性。与 UniProt 中记录的相比,Cas9 序列的数量增加了 4.1 倍、Cas12a 多了 6.7 倍、Cas13 多了 7.1 倍。
接下来,团队采用了一种分层训练策略。他们利用 CRISPR-Cas Atlas 微调了 ProGen2 模型,使模型掌握 Cas 蛋白功能相关的特定序列特征。经此过程生成的 Cas9 样蛋白,其多样性达到已知天然变体的 4.8 倍,对于 Cas13 和 Cas12a 等天然蛋白较少的家族,生成序列的多样性分别提升了 8.4 倍和 6.2 倍。

图 | 多种 Cas 蛋白家族的产生
尽管目前已有多种 CRISPR-Cas 蛋白被应用于基因组编辑,Cas9 仍是使用最广泛的工具酶。
为提高 Cas9 样序列的生成效率和准确性,研究人员专门用约 24 万条 Cas9 序列对模型进行了进一步微调。他们以不同天然 Cas9 序列片段作为蛋白质语言模型的生成提示,产生了 35 万条候选序列,并通过计算机筛选评估序列质量和 CRISPR 兼容性,最终选出 209 条序列在人类细胞中进行实验验证。
通过将 209 种核酸酶质粒与 SpCas9 sgRNA 质粒共转染 HEK293T 细胞,靶向三个已知靶位点之一。在所有三个靶位点中,编辑效率存在显著差异,其中部分 Cas9 样蛋白的活性达到或超越 SpCas9。
其中,最佳候选蛋白 PF-CAS-182 在靶位点展现出与 SpCas9 相当的编辑活性,将其命名为 OpenCRISPR-1。OpenCRISPR-1 全长1380个氨基酸残基,与 SpCas9 存在 403 个突变差异,与 CRISPR-Cas Atlas 中最接近的天然蛋白也有 182 个突变差异。
在所有测试浓度和 gRNA 条件下,OpenCRISPR-1 的靶向切割比例均显著高于SpCas9。关键的是,OpenCRISPR-1的脱靶位点完全包含于 SpCas9 脱靶谱系中,证明其不会产生新的切割模式。
将 OpenCRISPR-1 转化为靶链切口酶(D10A 突变),并与腺苷脱氨酶 ABE8.20 融合,构建碱基编辑器。在 HEK293T 细胞中测试,使用 sgRNA 靶向含有腺嘌呤的基因组位点,观察到 35%-60% 的 A 到 G 编辑效率,与 SpCas9 切口酶的 ABE8.20 系统相当,且未产生插入缺失突变。

图 | 产生的核酸酶在人类细胞中发挥基因编辑器的作用
研究人员还设计一个完全合成的碱基编辑器系统,包括脱氨酶结构域。测试了两种最活跃的合成脱氨酶(PF-DEAM-1 和 PF-DEAM-2),与 SpCas9 或 OpenCRISPR-1 切口酶融合后,显示出与 ABE8.20 相当的编辑效率。
此外,研究者也设计了针对这些蛋白的 sgRNA。测试后发现 31 种设计的 sgRNA 在编辑效率上优于 SpCas9 的 sgRNA,部分 sgRNA 表现出显著改进。OpenCRISPR-1 在使用设计的 sgRNA 和 SpCas9 的 sgRNA 时表现相似,显示出较高的编辑效率。
自发布以来,成千上万的学术和行业研究人员已从开发抗旱作物到药物研发等各个垂直领域访问了 OpenCRISPR-1。Profluent 计划将 CRISPR-Cas Atlas 开源。公司团队团队受到启发,正在向新的领域拓展,包括碱基编辑、Prime 编辑、大型插入技术等等。
参考链接:
1.Ruffolo, J.A., Nayfach, S., Gallagher, J. et al. Design of highly functional genome editors by modelling CRISPR–Cas sequences. Nature (2025). https://doi.org/10.1038/s41586-025-09298-z
免责声明:本文旨在传递合成生物学最新讯息,不代表平台立场,不构成任何投资意见和建议,以官方/公司公告为准。本文也不是治疗方案推荐,如需获得治疗方案指导,请前往正规医院就诊。























































































安各洛(深圳)生物科技有限公司 版权所有