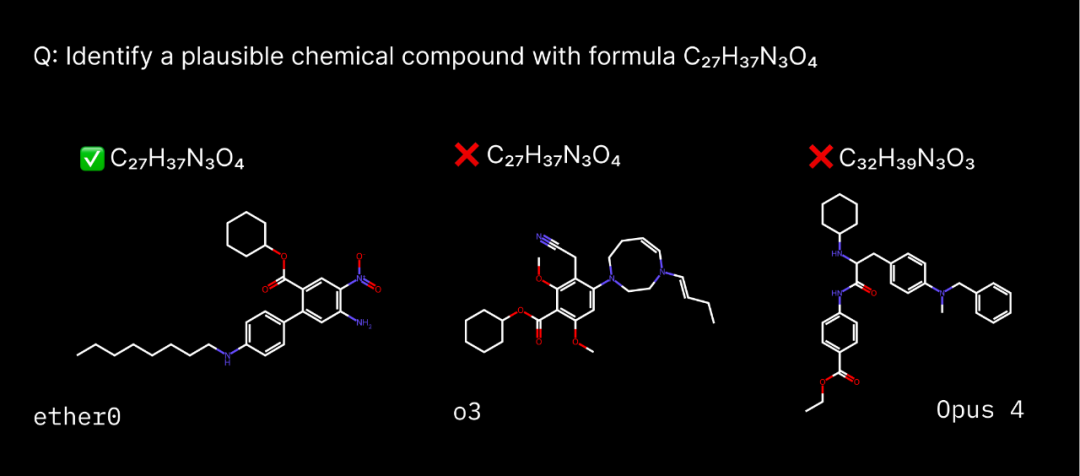
蛋白质结构预测一直是一个重大挑战性。在过去的 20 年里,这个领域的发展相对缓慢。然而,AlphaFold 的出现使得这个领域取得了迅速进展,预测准确性达到了或者接近了实验精确度。
近日,来自深圳湾实验室系统与物理生物学研究所的周耀旗团队在《国家科学评论》上发表题为“3=1+2: How the Divide Conquered de Novo Protein Structure Prediction and What’s Next?”的综述,梳理了近年来蛋白质预测领域的发展,并对 AlphaFold2 之后,该领域未来的方向做出展望。
作者们认为,AlphaFold2 带来的突破是建立在预测蛋白质一维结构特征和二维侧链距离方面的进展,以及通过一维和二维特征的嵌入来进行端到端的学习。AlphaFold2 把蛋白质结构预测这个三维结构问题拆解成一维和二维的子问题(即 3=1+2),解决了将多个同源序列映射到其关联的“单一”结构的问题,但却尚未能解决将单个序列映射到其真实世界的结构和动力学的问题。
技术背景
蛋白质在每个生物过程中都发挥着多种多样的作用,因此是分子生物学研究的焦点。蛋白质的化学结构简单,是由 20 种氨基酸残基的不同排列组成的线性高分子。蛋白质的功能多样性归因于它们能够折叠成独特而多样的三维结构形态。然而,通过实验确定这些蛋白质的结构费时费力且价格昂贵。
从 1994 年开始,结构预测技术的进展根据全局距离测试 GDT 打分函数来衡量。经过 20 年的努力,对于最难预测的蛋白质,总体的 GDT 分数只是从 27 增加到了 32。然而,DeepMind 开发的 AlphaFold 却在 2018 年让这个分数骤然增加到了 65 分,并在 2020 年通过 AlphaFold2 达到了 85 分,等同或者接近了实验测定的结构精确度。这一革命性的进展在生物学领域产生了深远影响。值得研究的是:这一飞跃是如何实现的,未来将如何进一步发展?
3=1+2,分而治之
由于存在巨大的构象空间和缺乏准确的自由能函数,从蛋白质序列直接预测三维结构挑战巨大。因此,这个问题长期以来被分成几个更简单的子问题,包括预测一维性质的二级结构或扭转角,以及二维性质的距离图,然后综合起来进行三维结构的预测。这种化繁为简的方法可以看作是一种“3=1+2”的方法。
一维结构特征的预测以预测三态二级结构为主。但是基于单一蛋白质序列的方法并不那么准确,即使是使用最先进的深度学习技术如 SPOT-1D-Single 和 S4PRED,三态准确性(螺旋,片条和无规线圈,Q3)也仅为 74%。而使用进化信息则能大幅度改进准确率。到 2019 年,作者们的 SPOT-1D 方法已经可以达到 87% 的准确率,接近理论极限。
不过用离散的二级结构状态来预测蛋白质主链的三维结构有其不足之处。例如,并不存在理想的螺旋和片条状结构,而所预测的无规线圈状不包含结构信息。相比之下,如果主链的三个扭转角(φ、ψ 和 ω)的数值已知,则可以直接构建主链结构。考虑到 ω 经常固定在 180°,作者们首次尝试预测两个扭转角(φ 和 ψ)的实数值。其中 Real- SPINE 预测的 ψ 角起始误差平均为 54°。通过解决角度的周期性问题(RealSPINE2)、改进神经网络、离散和连续实数值的混合预测(SPINE XI),并结合最新的基于深度学习的多种方法,误差最终被降低到 20°。
回顾主链结构预测从离散到连续的演变,可以发现这个过程催生了使用针对特定蛋白质的能量函数来进行无结构碎片蛋白质结构预测的方法,以及通过可微分损失函数来实现端到端结构预测的方法。
而对于二维结构特征,则需要预测 Cα 原子或侧链 Cβ 原子的二维离散接触或连续距离图。与主链结构预测类似,2016 年左右通过将深度学习与直接耦合分析(如 PconsC2 和 RaptorXContact)相结合,接触预测的精确度得到了很大的改进。随着深度学习的不断发展,离散两态接触图的预测进一步发展为距离网格图甚至连续距离预测。就像主链结构的预测从离散转变为连续一样,从离散到连续的距离预测则为“3=1+2”方法在连续可微空间中的应用铺平了道路。
三维结构预测及AlphaFold
在 AlphaFold 出现之前,蛋白质结构预测始于同源建模或模板建模。由于大多数蛋白质无模板,因此科学家们开发了无模板建模,即 CASP 自由建模,也称从头结构预测。Bowie-Eisenberg 首次提出基于结构碎片的技术,将序列片段映射到结构碎片,进行能量引导的结构组装。使用刚性结构碎片可更快采样,缺点是很难定位最佳结构碎片,而且天然结构碎片与结构库里面的最佳结构碎片之间或许不能完全吻合。
更自然的蛋白质折叠方法是无模板/无结构碎片建模。科学家们开发了基于知识的结构碎片方法,采用扭转角采样偏差或预测的扭转角作为针对蛋白质的评分/约束。然而,尽管能够准确预测扭转角,且对于生成主链结构很有帮助,这些新出现的无结构碎片方法被在 CASP 上发展比较成熟的基于片段的方法边缘化了。
在 CASP13 中,AlphaFold 使用的也是无片段的蛋白质特异性的评分方法。它是建立在前人方法的基础上的。例如 CONFOLD 和 COINFOLD 利用了预测的离散的二级结构和接触图 。而作者提出的方法 SPOT-Fold 采用了预测的连续角度,但具有离散的预测接触图。相比之下,AlphaFold 预测了残基之间的距离和主链扭转角的分布,并将两者转换为一个蛋白质特异的评分函数以进行最小化。AlphaFold 表明,“在 CASP 中后来使用的无结构碎片梯度下降方法在每个类别中的表现都优于结构碎片组装方法”,这证实了无结构碎片方法与特定蛋白质的能量函数相结合的优势。AlphaFold 将科学界的注意力引向了无结构碎片技术,并利用蛋白质特异的评分,在几乎连续的网格空间中,利用了 3=1+2 的方法来实现了蛋白质结构预测精度的第一次飞跃。
AlphaFold2 的端到端学习
AlphaFold2 在 CASP14 中进一步推动了无结构碎片的方法,但不再使用能量函数,而是通过神经网络进行序列到结构的直接映射。AlphaFold2 输入同源序列以提取一维和二维演化特征,并直接预测全局仿射变换和局部侧链扭转角以促进全原子结构组装。其所预测的结构在结构模块和模型循环中更新,这与之前的 AlphaFold 通过“1”和“2”以产生“3”的方法不同。
AlphaFold2 的网络架构来自于计算机科学中可微分反向传播技术的发展,这使得端到端的蛋白质结构预测成为可能。RGN 和 NEMO 是早期尝试端到端蛋白质结构预测的模型,它们通过迭代优化和主链角度预测并强制执行链约束。AlphaFold2 借鉴了 NEMO 的方法,但做出了两个关键改进:通过预测独立的残基仿射变换生成三级结构,并从多个同源序列映射到单个结构的角度出发进行预测。这样可以将多个同源序列一起输入,从而可以更直接地捕获协同进化信息,这与 RGN 和 NEMO 中使用的预处理一维序列谱不同,因为那样很可能丢失了大部分协同进化信息。此外,AlphaFold2 采用了庞大的序列数据集(BFD)和 PDB 中的大多数结构(170,000个)来训练一个大模型。训练这个大模型所需的计算资源是大多数科研团队无法获得的。
在 AlphaFold2 之后,科学家们还陆续开发出了许多端到端方法,包括 RoseTTAFold、RGN2、ESMFold 和 OmegaFold。大多数新方法都侧重于使用语言模型进行结构预测。语言模型包含了进化信息,不需要进行同源序列搜索,可以加速推理速度,但该类方法通常会伴随着建模精度的下降,对于较小的蛋白质家族尤甚。
展望未来
作者们认为 AlphaFold2 仅仅解决了将多个同源序列映射到单个结构的问题,因此它仅对具有大量同源序列的蛋白质能够预测准确的结构。使用蛋白质语言模型不是解决方案,因为语言模型从多个密切相关的序列中隐式地学习了同源信息。依赖同源序列意味着 AlphaFold2 无法预测少量突变所引发的有害的结构变异。最终的挑战是确定一个序列与其结构之间的对应关系,但是距离解决这个问题还有很长的路要走,因为基于单个序列预测二级结构的准确性约为 74%,远低于理论极限值 86-90%。要提高预测能力,首先必须在单个序列水平上将二级结构的预测准确性提高到 86%。
而预测蛋白质动态、构象转变、突变引起的蛋白质稳定性变化和蛋白质-配体相互作用将更具挑战性,因为这些预测更要考虑单个序列特异性,所以不同的同源序列可能有不同的表现。不过,作者们预计在不久的将来,蛋白质-蛋白质复合物结构预测和蛋白质设计将取得重大进展。其中,AI 驱动的蛋白质设计逐渐成为主流,且已从固定骨架设计发展到结构序列生成器。预测蛋白质-蛋白质复合物结构可能很快会更好利用协同进化信息。
RNA 结构预测是另一个相关领域,但在 CASP15 评估中,基于 BRiQ 统计能量函数的传统能量方法表现最佳,而大多数 AI 方法表现不佳。AI 驱动的 RNA 结构预测面临的挑战包括信息有限、缺乏序列保守性、主链构象角度多以及 RNA 结构在蛋白质数据库中稀少。尽管如此,在 RNA 全碱基配对结构方面还是取得了一些进展,使用了 SPOT-RNA、SPOT-RNA2、RNAcontact 和 SPOT-RNA-2D 等方法。随着更多数据和更好的算法的出现,AI 驱动的 RNA 结构预测肯定会在不久的将来重新成为焦点。
因此作者认为,AlphaFold2 在将同源序列映射到单个结构上的成功并不是结构生物学的终结,而是一个 AI 与实验结合新时代的开始。






















































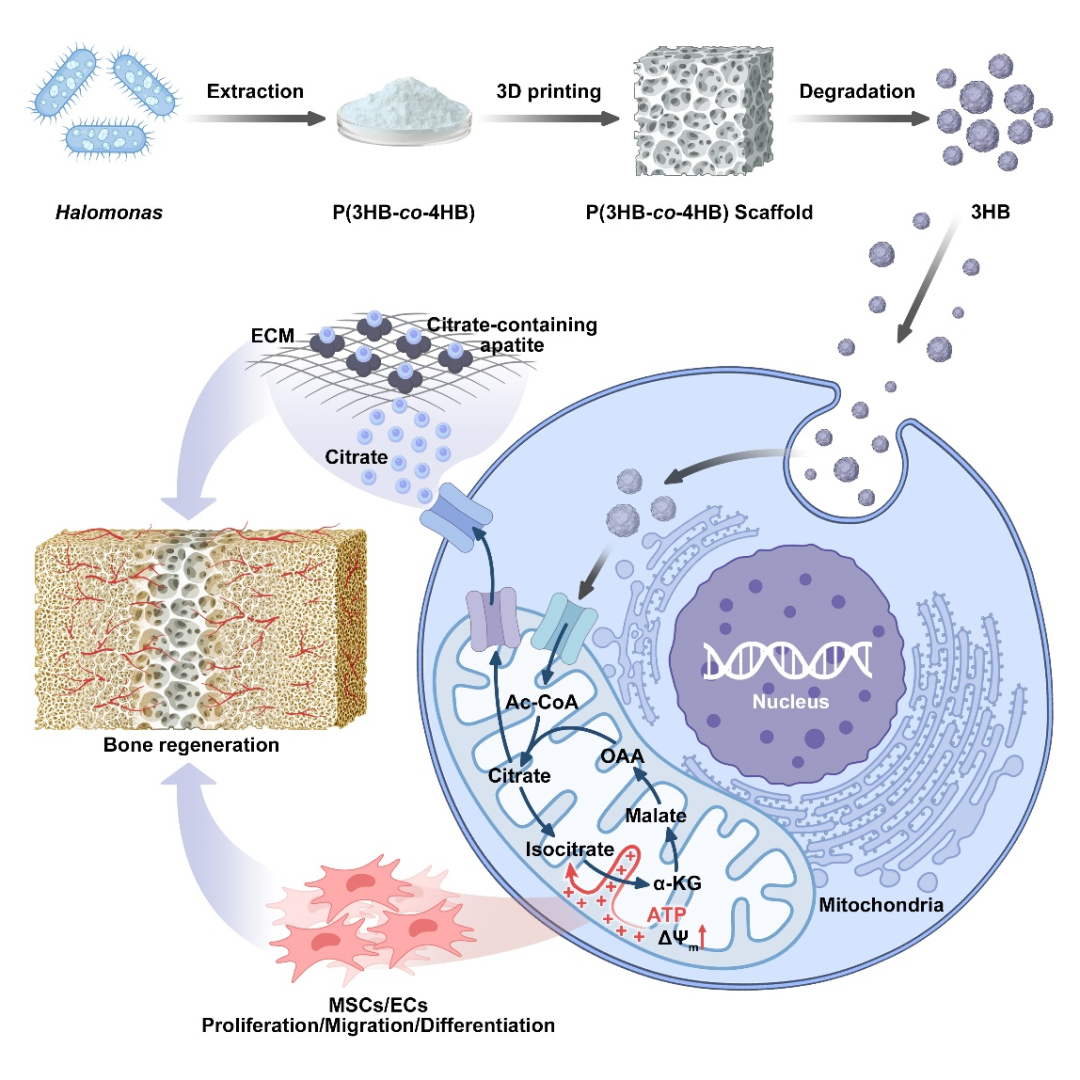
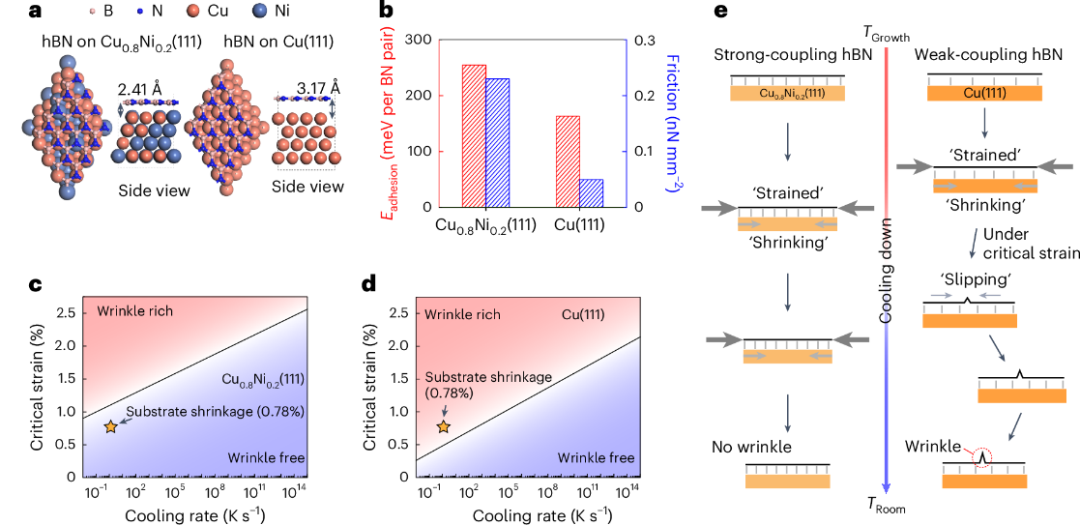








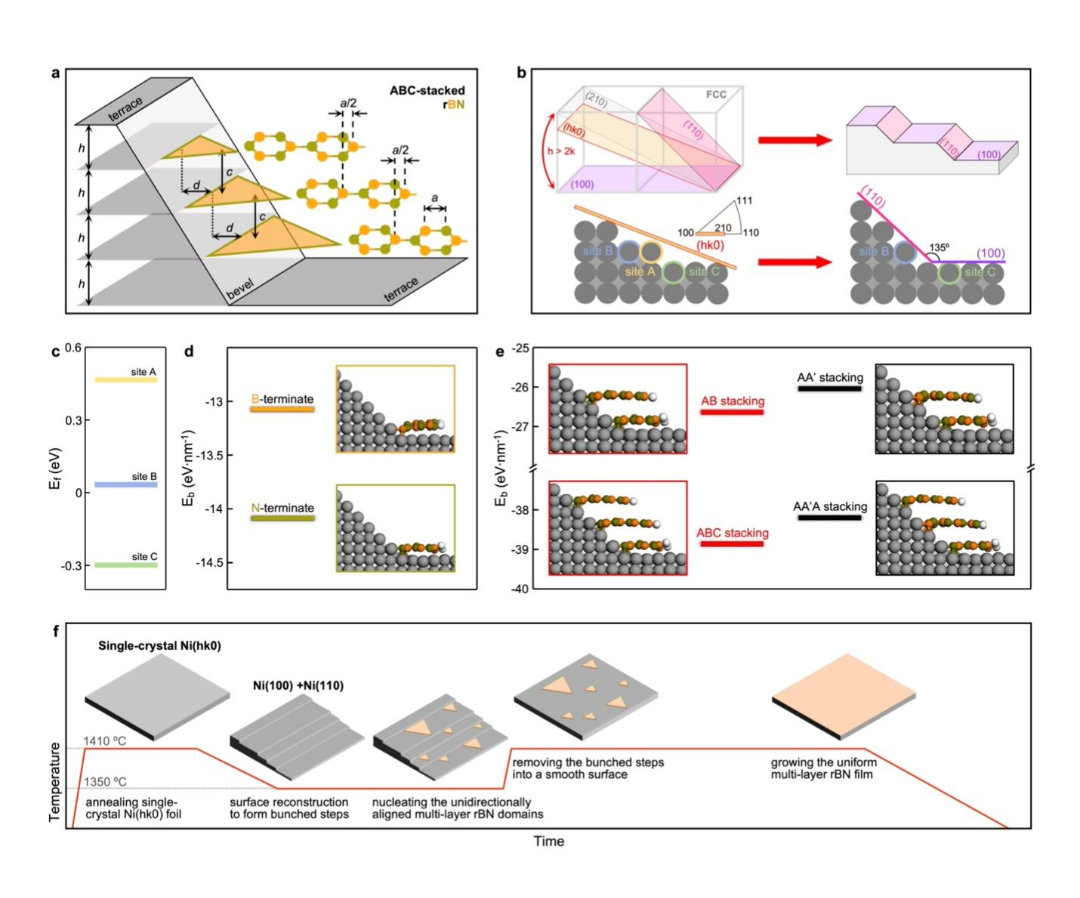

























安各洛(深圳)生物科技有限公司 版权所有