
“在这项研究中,我们第一次在细胞水平上将光合系统完整的超大蛋白质复合物的原位结构解析,推进到 3.3 埃(angstrom,Å)的近原子分辨率水平。”中国科学院院士、清华大学隋森芳教授表示。

图丨隋森芳(来源:隋森芳)
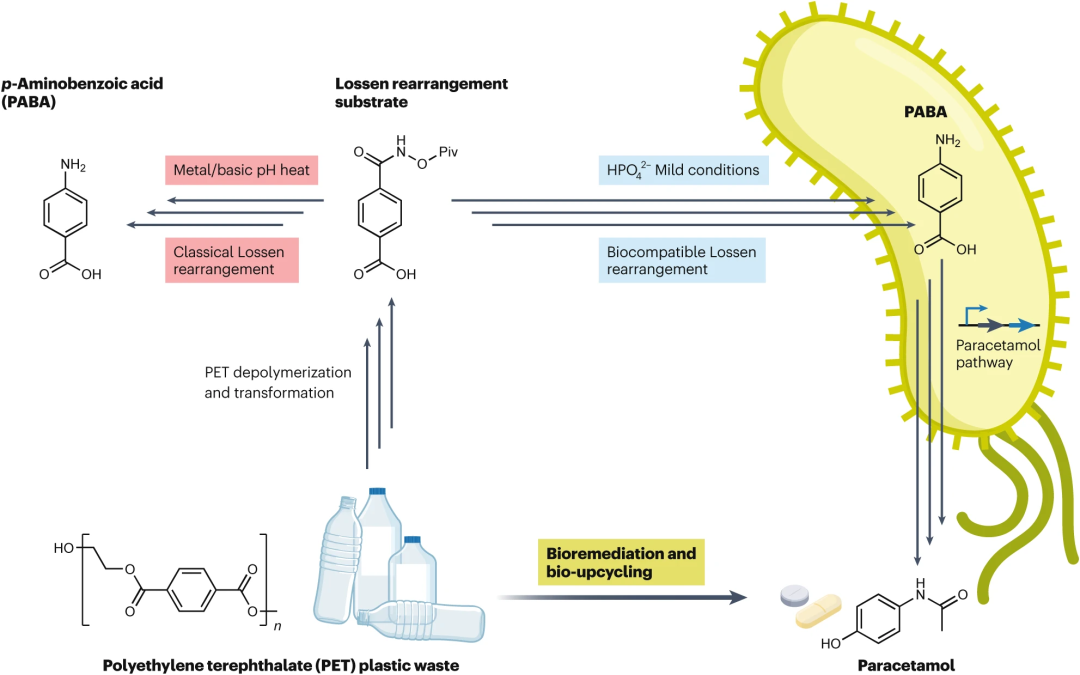
我们都知道,氧气乃生命之源。而光合作用,即绿色植物通过吸收光能,将水和二氧化碳转化为有机物,并释放氧气的过程,则是地球上氧气的主要来源。
在细胞内部的光合系统中,太阳光先被天线系统捕获,然后传输到类囊体膜上的光系统 II(photosystem II,PSII)和光系统 I(photosystem I,PSI),从而诱导随后的电荷分离,以驱动光合作用的发生。以红藻为例,其捕光天线系统包含水溶性藻胆体(phycobilisomes,PBS)和跨膜捕光复合物(light-harvesting complexes,LHC)。
目前,由于缺乏对完整的光合系统结构的高分辨率解析,相关领域的科学家们对于 PBS 捕获的能量究竟是如何传输到光系统 II 和光系统 I,以及能量在光系统 II 和光系统 I 之间的分配情况尚不明晰。
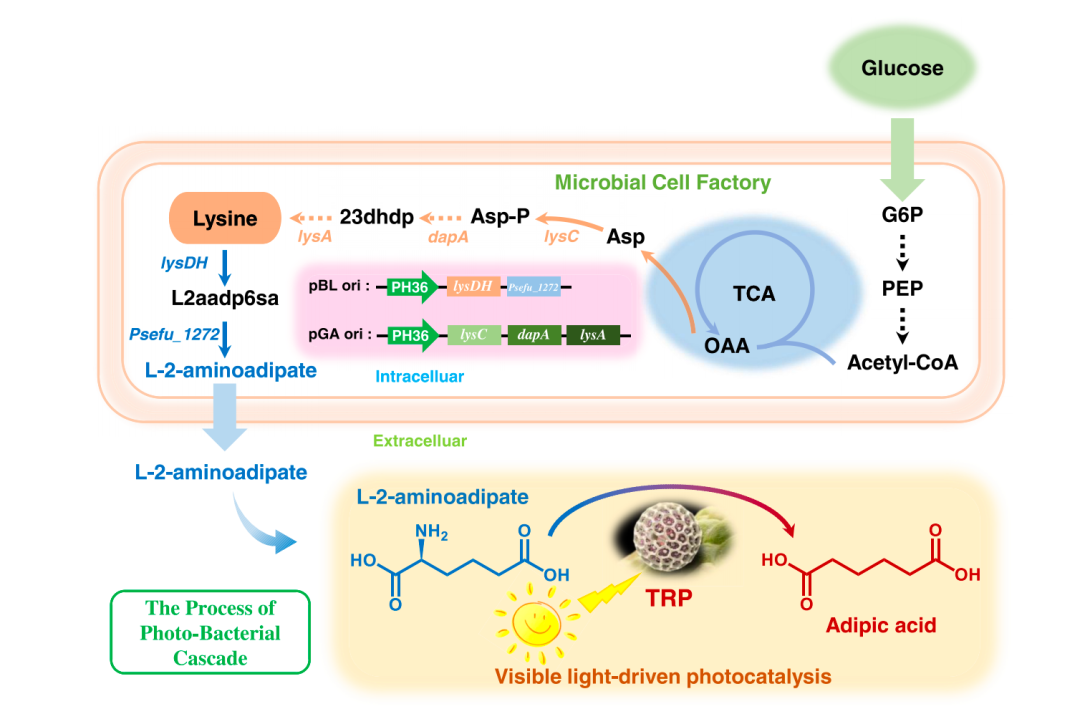
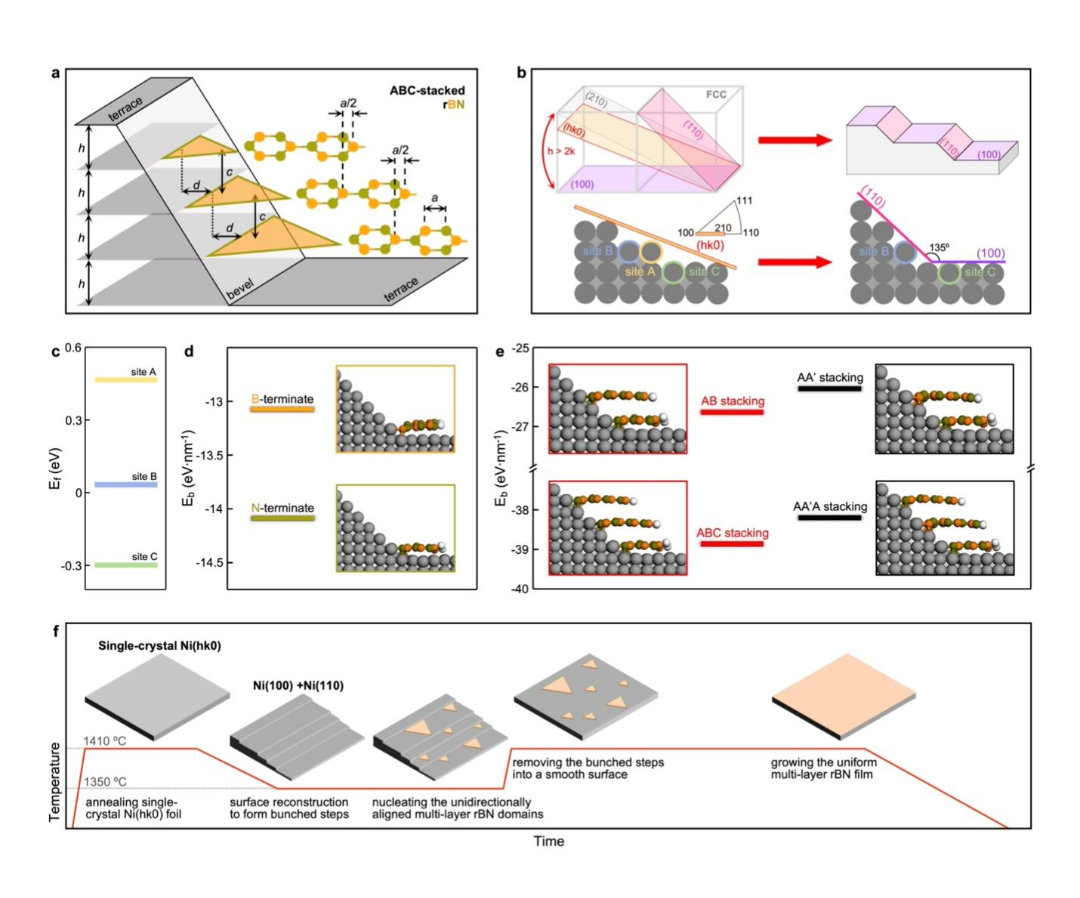
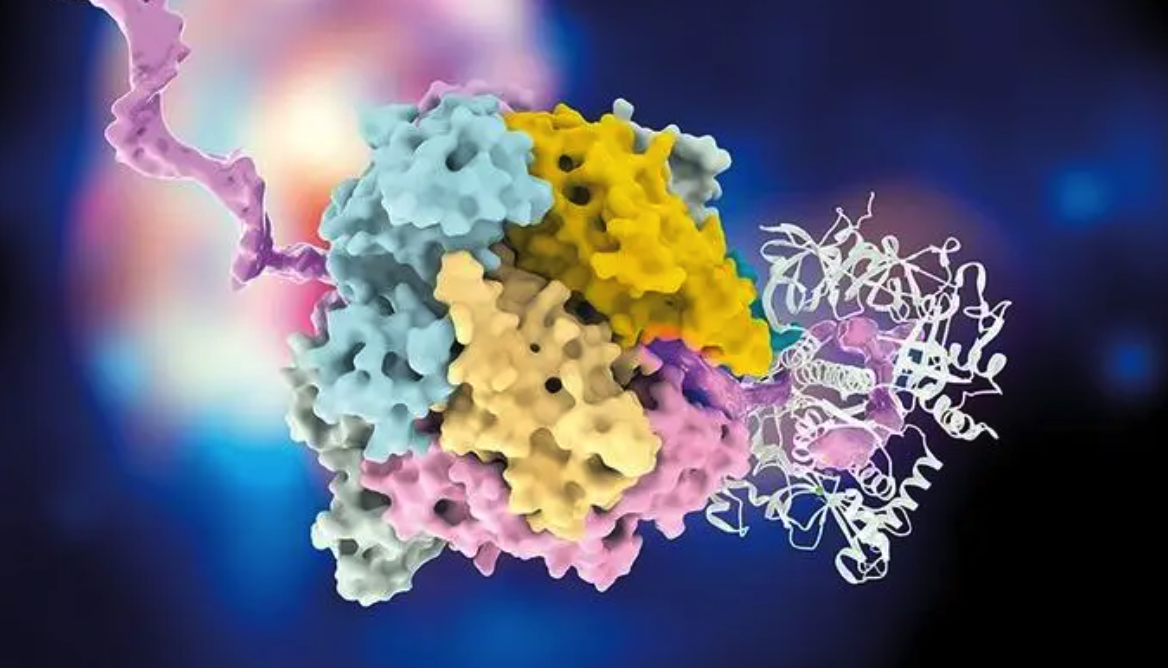
为了解决上述问题,清华大学隋森芳院士团队、王宏伟教授团队以及中国科学院生物物理研究所章新政研究员团队联合,把冷冻电子断层技术(cryo-electron tomography,cryo-ET)和原位单颗粒技术(in situ Single particle analysis, isSPA)相结合,解析了盐泽红藻 PBS -PSII-PSI-LHC 超大复合物的原位结构,将单 PBS-PSII-PSI-LHC 和双 PBS -PSII-PSI-LHC 两者的总体分辨率分别推进至 3.3Å 和 4.3Å,并提供 PBS、PSII 和 PSI 之间相互作用的详细信息。
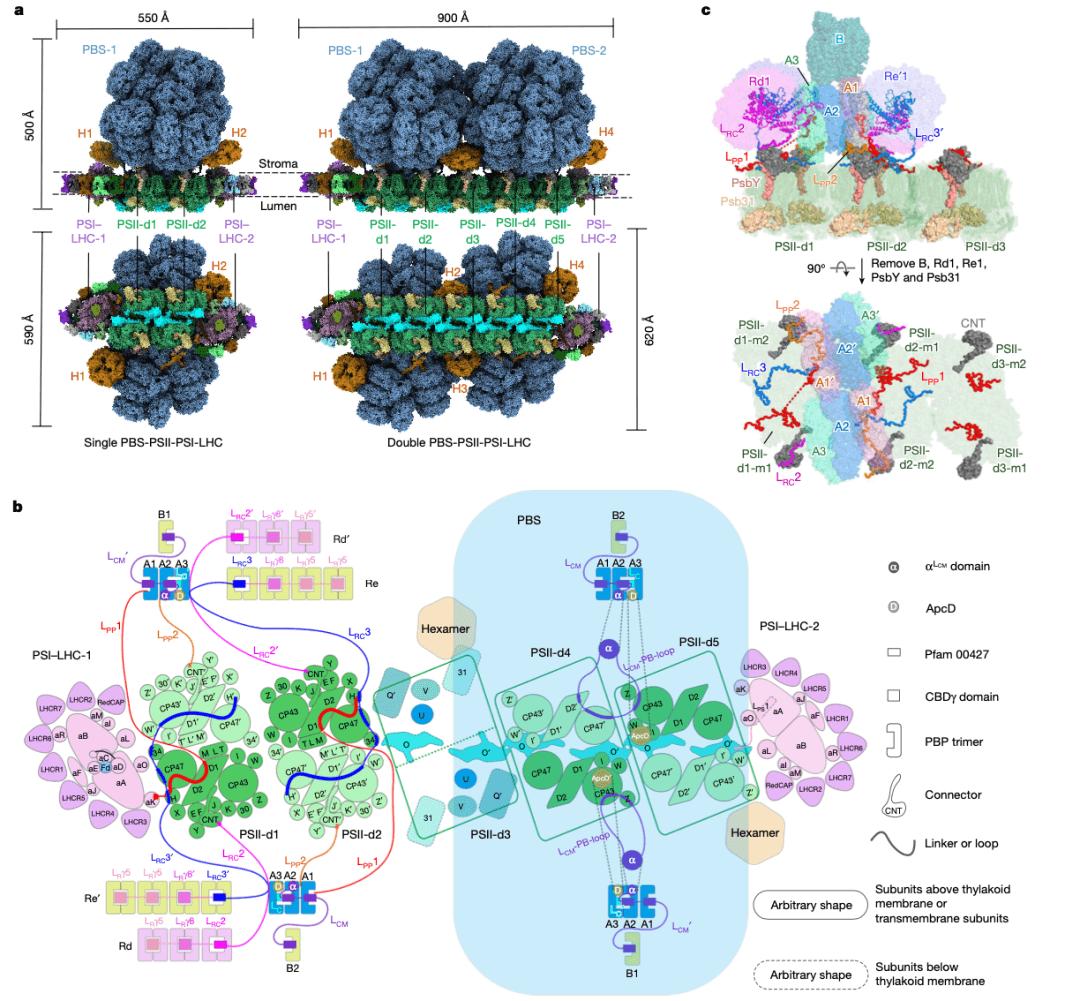
图丨盐泽红藻 PBS -PSII-PSI-LHC 超大复合物的原位结构(来源:Nature)
近日,相关论文以《红藻藻胆体-光系统 II-光系统 I-捕光复合物超大复合体的原位结构》(In situ structure of the red algal phycobilisome–PSII–PSI–LHC megacomplex)为题在 Nature 上发表[1]。

图丨相关论文(来源:Nature)
清华大学 2019 级博士研究生游鑫、博士后张星、中国科学院生物物理研究所博士后程静和南方科技大学博士后肖亚男担任共同一作,清华大学隋森芳教授、王宏伟教授和中国科学院生物物理研究所章新政研究员担任共同通讯作者。
论文审稿人表示:“这是一项开创性的工作,代表了一种通过结合不同的成像方法,以研究大尺度颗粒的样品的新道路。”

实现近原子分辨率水平的超大复合物原位结构解析
细胞活动的基本问题,实际上是生物大分子之间相互作用的问题。要回答这一问题,首先需要厘清其中的结构信息。因此,以研究生物大分子的三维空间结构、动态过程及生物学功能为主的结构生物学,可以被看作生命科学的基础。
作为结构生物学的主流技术,单颗粒冷冻电镜技术如今已经能够把生物大分子的结构解析,推进到接近 1Å 的原子分辨率水平,并分辨出如络氨酸侧链残基苯环上的氢氧原子。
不过,在使用该技术之前,必须先将样品蛋白进行分离纯化,才能够进一步收集数据。另外,结构解析要求颗粒的全同性,以便通过平均提高分辨率。因而,如果想对细胞或组织这样的原位样品进行结构解析,这种技术并不实用。
冷冻电子断层扫描技术(cryo-electron tomography,cryo-ET),是目前解析细胞或组织原位结构的常用方法,先利用冷冻电子显微镜收集三维断层图像数据,再使用子断层图像平均法(sub-tomogram averaging,STA)解析结构。
该方法与单颗粒冷冻电镜技术有所不同,其主要用来研究不具备全同性的细胞结构,并需要通过倾转收集样品数据。这里所说的倾转,是在保持入射电子束不动的基础上旋转样品台,从不同的角度采集图像数据,再通过图像的重构,得到样品的三维空间结构。
但是,即便采用现下最好的硬件设备,基于 cryo-ET 得到的分辨率,一般只能达到 20Å 左右,无法解决细胞或组织内结构在分子水平上的相互作用问题。
“为了进一步提高 cryo-ET 的分辨率水平,我们通常选择把单颗粒的方法整合到 cryo-ET 中。比如,我们可以用 cryo-ET 方法,解析出数以百计的线粒体结构,再用 STA 方法从中挖出成千上万个线粒体中核糖体的颗粒,并对颗粒进行平均,这样便能很好地提升核糖体颗粒密度的分辨率。”隋森芳表示。
这是目前通用的原位结构解析流程,即 cryo-ET-STA, 正是采用上述方法,隋森芳团队于 2021 年成功地解析了一个原位的光合系统结构[2]。虽然在该结构中,他们已经能够实现 15Å 左右的高分辨率水平,但还无法揭示分子之间相互作用的细节信息。
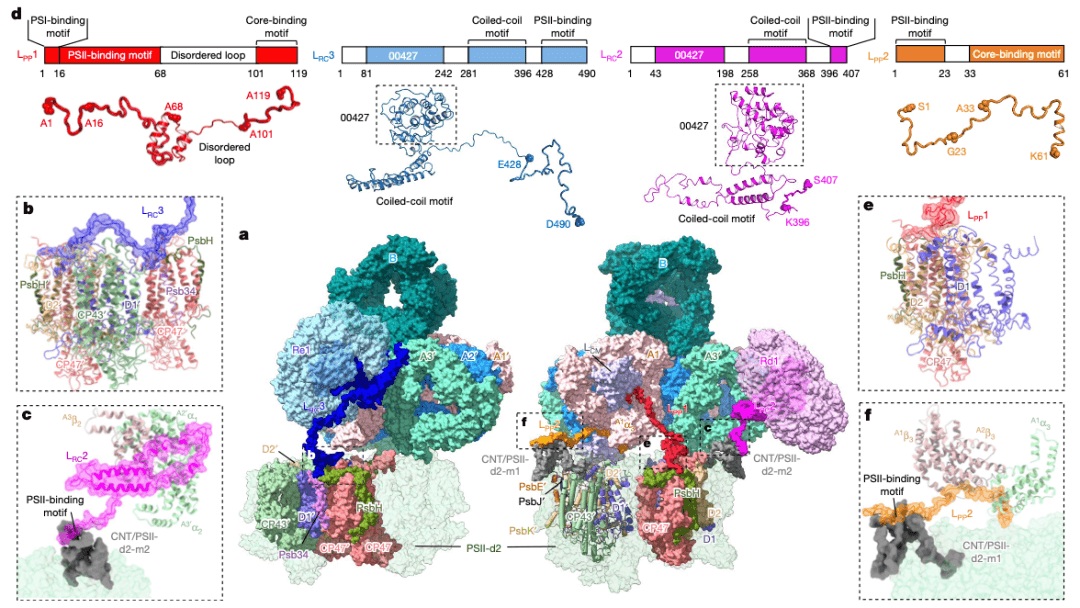
图丨PBS 和 PSII 通过四个连接蛋白连接(来源:Nature)
“这时候我开始意识到,用传统方法收集数据、解析结构,不能实现我们想要达到的目标。所以,我们必须另辟新的办法。”隋森芳表示。
他带领团队查阅了大量文献,并与王宏伟课题组和章新政课题组开展合作,最终决定将 cryo-ET-STA 和 isSPA 这两种方法结合起来,对目标生物样品进行原位结构解析。
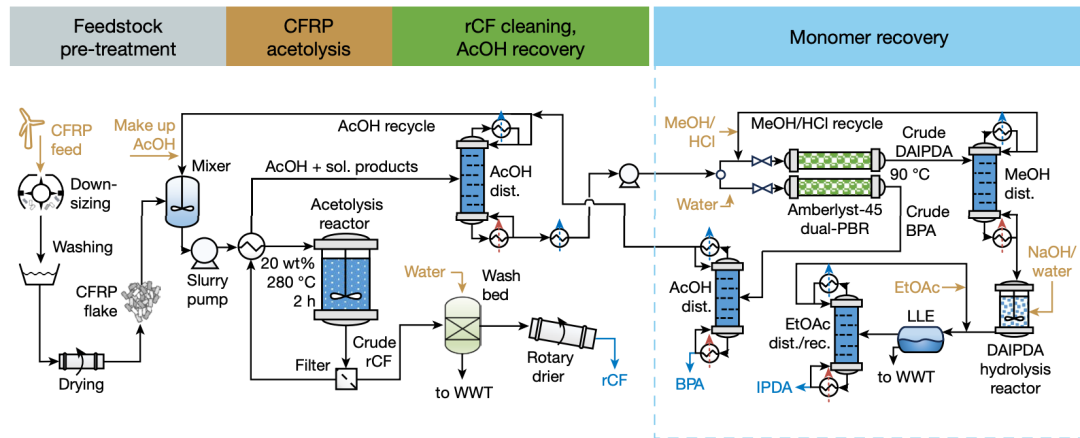
具体来说,把细胞切片样品分成两组,其中一组用 cryo-ET 采集倾转的图像数据,另一组用单颗粒方法采集非倾转图像数据。基于前者,他们通过 cryo-ET-STA 处理流程得到了分辨率水平为 8Å 的光合系统超大复合物结构,并将该结构作为模板,在另一组非倾转的细胞切片数据中挑选出光合系统超大复合物颗粒,然后用原位单颗粒方法进行结构的精细计算。
隋森芳表示:“最终,我们成功地在原位条件下把超大蛋白质复合物的结构分辨率推进至 3.3Å。其中,光合系统复合物中的 PBS 核心部分,已经达到 3Å,这是迄今为止细胞切片样品所能达到的最高分辨率,标志着我们完全能够在细胞原位条件以近原子分辨率量级,阐释生物大分子之间的相互作用。”

“老路子行不通时,就要寻找新路子”
隋森芳介绍,对光合系统的结构进行研究,其实验室已拥有十多年的历史。
2010 年之前,因为技术条件比较差,样品也很大,因此解析出的 PBS 结构,普遍在 20Å 至 30Å 的范围内。
2014 年前后,直接电子探测器诞生,冷冻电子显微镜技术迎来分辨率革命。基于新的电镜技术,该团队采用单颗粒方法,先于 2017 年报道了国际上第一个分辨率为 3.5Å 的 PBS 结构[3],解决了 PBS 结构如何组装的问题。2020 年,他们将 PBS 结构的分辨率推进至 2.8Å 的水平,揭示了 PBS 内部能量传递的调节机制[4]。基于这些成果,他们得以在近原子水平上实现 PBS 结构解析。
不过,如上所说,除了 PBS,光合系统还包括类囊体膜上的 PSII 和 PSI。因此,必须解析出一个完整的光合系统结构,才能解决 PBS 的能量传输机制和分配机制。
“传统的分离纯化方法并不能解决这一问题,因为捕光天线 PBS 是水溶性的,而膜蛋白 PSII 和 PSI 是脂溶性的,要想从生物化学角度将其完整地分离出来几乎不可能。所以,只能采用原位的细胞结构解析方法,但通常的原位结构解析方法分辨率水平上不去,还无法实现最终目标。”隋森芳解释道。
“老路子行不通的时候,就要去寻找一条新的道路。”他说。在探索新方法的过程中,该团队也经历过一段彷徨的时期,最终经过不断优化和整合方法,通过 cryo-ET 与 isSPA 的结合实现了 3.3Å 分辨率的光合系统超大蛋白复合物的结构解析。
“总的来说,我们关于光合系统的研究工作,经历了分辨率从 20Å 至 30Å 的纳米水平,再到 3Å 的近原子水平。从技术上看,我们走过了两个技术环节,从用单颗粒方法到用电子断层成像与原位单颗粒结合的方法来解析结构。从样品上看,我们则经历了从基于离体的分离纯化样品来解析结构,到在原位细胞内部直接解析结构的过程。”隋森芳表示。
那么,研究光合系统,对应用层面都有哪些促进呢?
一方面,厘清光合系统的能量传递路径,有助于助力“双碳”目标实现。本项研究成果所涉及的光合系统,是一种海洋藻类,也是海洋浮游生物的重要组成部分,而地球上每年所排放的二氧化碳,有一半都被后者吸收。
另一方面,有利于保护海洋生物、维护海洋生态及地球上的“固碳”功能。比如,对于主要通过光合蛋白质吸收能量的海洋生物来说,其在遭受破坏时,首先被破坏的便是能量获取机制,所以该成果能促进光保护机制等研究。
此外,由于在本项研究中,包括色素、叶绿素等在内的光合系统结构已经能够被非常清晰地确定。因此,对于利用人工方法开发新的光合元件具有指导意义。
据了解,目前,该团队正在将 cryo-ET 和 isSPA 结合的新方法,推广到其他的研究对象中,希望能够助力解决更多科学问题。
运营/排版:何晨龙























































































安各洛(深圳)生物科技有限公司 版权所有