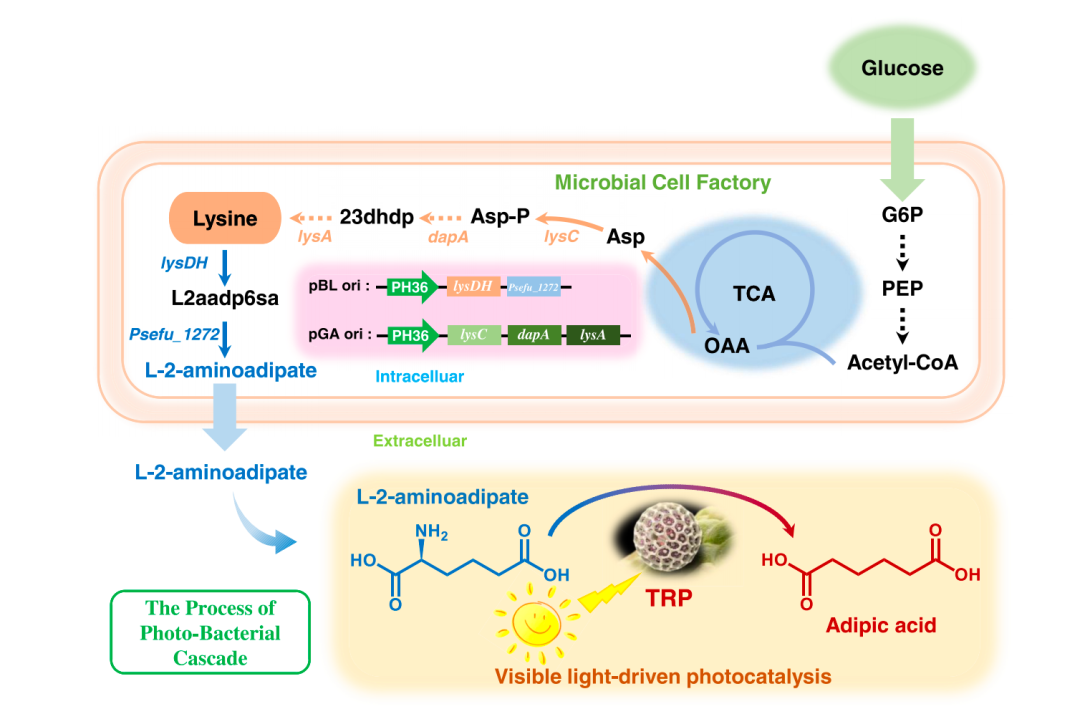
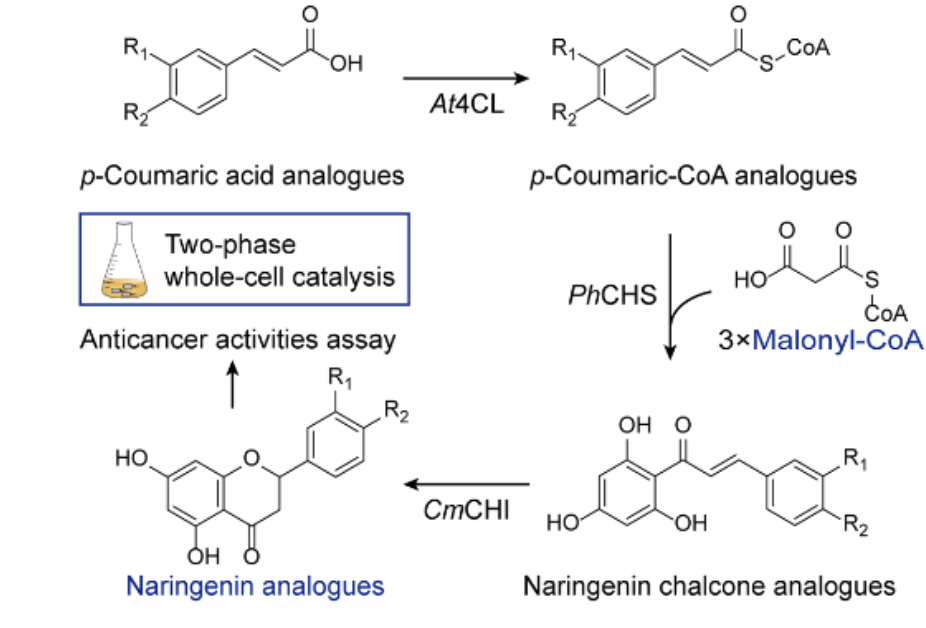
随着信息技术的发展,数据真正成为了一种新型生产资料。不过,它的爆炸式增长也让存储成为新的挑战,基于硅材料的计算机存储方式逐渐变得难以应对。在此背景下,积极探索 DNA 存储以作为下一代的数据存储方式意义重大。
DNA 是一种非常稳定的分子,半衰期超过 500 年,低温条件下可保存成千上万年。DNA 存储在存储密度、寿命和能耗方面已显示出超越当前硅基数据存储技术的潜力。
DNA 存储的整个流程可大致分为 4 个步骤,即“编”“写”“存”“读”。如今使用 DNA 测序仪读取 DNA 四个字母的编码数据非常简单,而且速度相对较快。问题在于写入数据,这通常需要一次合成一个字母的定制 DNA 链。当今最快的 DNA 写入器每天可以合成约 3.2 亿字节的 DNA 数据,按照这种速度,写入 1g DNA 需要近 200 万年。且由于写入速度慢,成本也较高。
近日,北京大学定量生物学中心钱珑、欧阳颀,北京大学计算机学院张成,亚利桑那州立大学Hao Yan 等人在 Nature 发表最新研究成果,他们受表观遗传修饰和 DNA 自组装启发,开发了一种非常规的 DNA 数据写入框架,该框架允许基于 DNA 自组装引导的酶促甲基化将任意的“表观比特”(epi-bits)以并行方式稳定地写入 DNA 模板上。
这种方式不用从头构建 DNA,而是利用现有的 DNA 链,在合成后对其进行修饰。“epi-bits”的功能类似于活字印刷术中的活字,可以排列在通用的 DNA 模板上。
基于此方法,他们成功将中国古代汉代的老虎拓片图像和熊猫彩色图片存储在 DNA 中,并成功读出。文章题为“Parallel molecular data storage by printing epigenetic bits on DNA”。
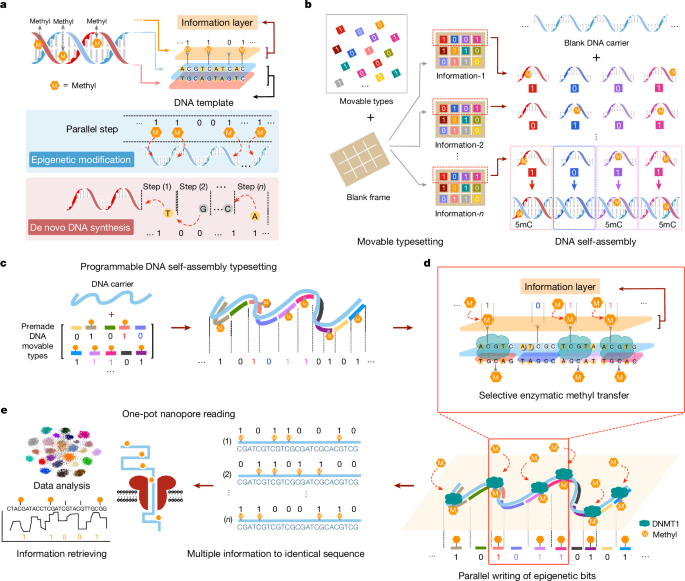
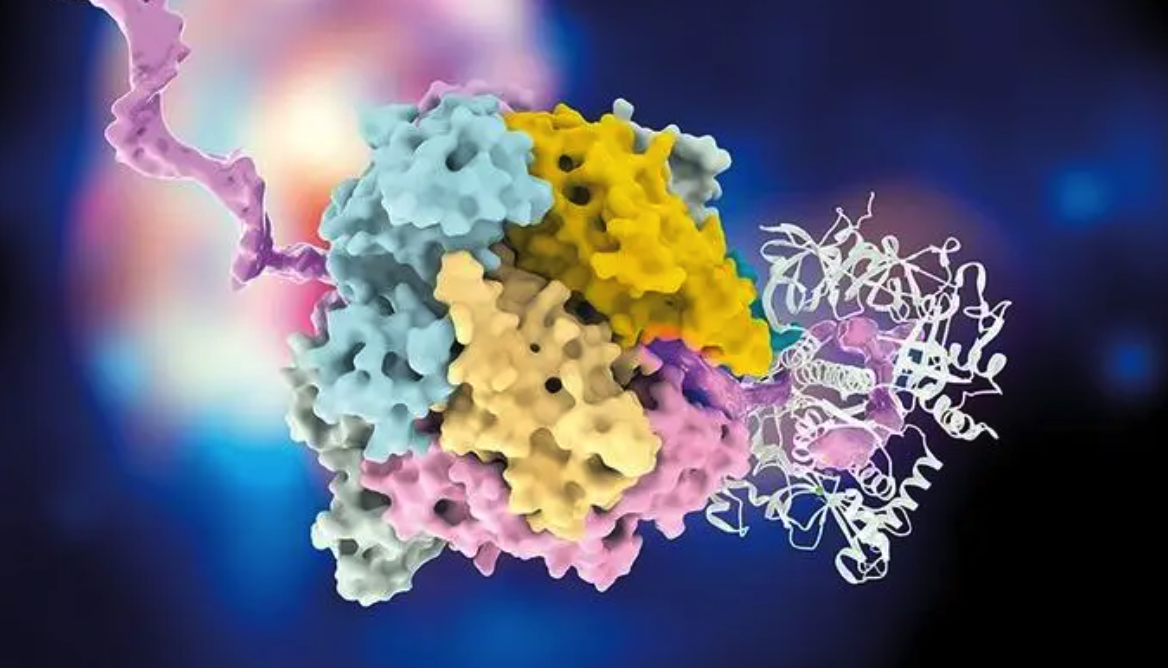
图 | epi-bit DNA 存储示意图
他们合成了标准化的长模板单链 DNA 片段作为“纸张”,一系列短的、预制的 DNA 片段(有或没有甲基)作为“字体”,这些 DNA 片段每个长 24 个碱基,其序列被设计为与 DNA 模板上的特定区域结合。
利用甲基化,用数字 0 或 1 对短 DNA 片段进行编码,甲基的存在对应 1,甲基的缺失对应 0。在体内,细胞将甲基附着到特定的 DNA 序列上,以发出信号,指示哪些基因应该在不同组织中表达和沉默。
研究人员添加了甲基转移酶 DNMT1,通过 DNMT1 的选择性甲基化,表观遗传修饰作为信息位可以精确地引入到通用 DNA 模板上以实现分子活字印刷。
基于上述方法,研究人员存储了 269,337 比特的数据,包括中国古代汉代的老虎拓片图像和熊猫彩色图片,实现了每次反应 350 比特的速率。
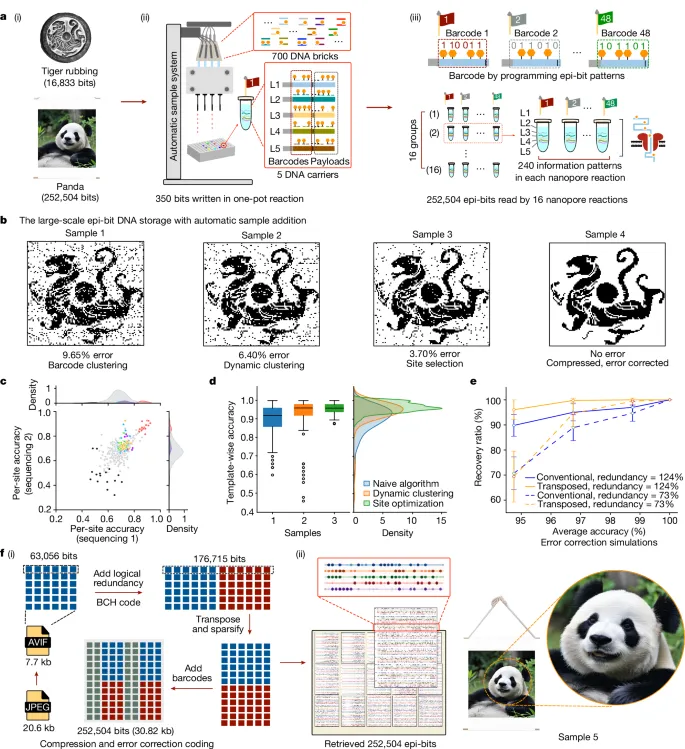
图 | 通过 epi-bit 实现大规模存储
此外,研究人员还邀请了 60 名具有不同学术背景的学生志愿者,在教室中使用简单的 epi-bit 书写工具手动将他们选择的文本片段存储在 DNA 中。最后,存储的 15 篇文本中有 12 篇被成功恢复,并通过在线服务器安全地返回给了编写它们的团队。
由于该技术使用预制的 DNA 片段,因此可以进一步优化以进行批量生产。这将比为存储的每一位信息合成一条定制的 DNA 链便宜得多。目前用这种方法写入数据的成本约为每比特 0.003 美元。但团队相信随着商业化的推广和应用,成本会降低。
下一步,团队将研究该系统的扩展能力如何适应大量数据。他们正在研究用甲基以外的其他化学标记修饰 DNA 模板,以便在每条链上编码更多的数据,从而进一步加快速度。
参考链接:
1.Zhang, C., Wu, R., Sun, F. et al. Parallel molecular data storage by printing epigenetic bits on DNA. Nature 634, 824–832 (2024). https://doi.org/10.1038/s41586-024-08040-5
免责声明:本文旨在传递合成生物学最新讯息,不代表平台立场,不构成任何投资意见和建议,以官方/公司公告为准。本文也不是治疗方案推荐,如需获得治疗方案指导,请前往正规医院就诊。























































































安各洛(深圳)生物科技有限公司 版权所有